Projet TOFoo

L’agriculture biologique est un des marchés les plus dynamiques du secteur agro-alimentaire (9,7 milliards d'euros en France en 2018, +15,7 % par rapport à 2017). C’est également une filière en mutation accélérée. Elle devient un secteur mondialisé aux échanges commerciaux intercontinentaux. Selon le besoin exprimé par les consommateurs, elle va aussi devoir passer progressivement de l’actuelle obligation de moyens à une obligation de résultats en termes de qualité et de sécurité des produits.
Pour vérifier le respect de son cahier des charges, la filière de l'agriculture biologique a mis en place un système de contrôle exemplaire dans le domaine agricole, fondé sur une réglementation européenne unique et un système de certification des opérateurs.
Annonce officielle de lancement du Projet TOFoo
Pourtant, elle reste vulnérable à des pratiques qui remettent en cause l’intégrité et la conformité des produits. En effet, la demande est structurellement excédentaire par rapport à l'offre. De plus, les chaînes logistiques se sont allongées, ce qui rend la traçabilité des lots plus complexe à vérifier. Surtout, les gains potentiels en cas de fraude sont substantiels du fait de prix de ventes plus élevés que dans le conventionnel. Or, les moyens actuels de contrôle ont des limites, que ce soit la traçabilité, la certification ou les analyses physico-chimiques. Cette vulnérabilité aux problématiques d'intégrité alimentaire commence à être perçue par les consommateurs, dont plus de 60 % ont des doutes sur le fait que les produits bio le soient réellement. La préservation de la confiance des consommateurs est ainsi devenue un enjeu majeur pour les prochaines années. L'ensemble des acteurs a besoin de démontrer que les produits biologiques sont authentiques et respectent réellement le cahier des charges bio. Le cœur du projet TOFoo est de développer un nouveau service d'authenticité des aliments afin de vérifier que le mode de production du produit testé est bien biologique. Ce service sera fondé sur une stratégie analytique dite non-ciblée.
La première innovation portée par le projet TOFoo consistera à mettre sur le marché un service qui aujourd’hui n’existe pas : l’authentification des produits alimentaires issus de l’agriculture biologique. Cette innovation, qui sera réalisée en mettant au point une méthode d’analyse multi techniques avec une stratégie non-ciblée, va radicalement changer la manière dont ces produits pourront être contrôlés. Cette innovation reposera sur plusieurs autres innovations technologiques, qui devront surmonter des difficultés technologiques propres.
La liste des partenaires sont :
Structure du projet et attribution des tâches
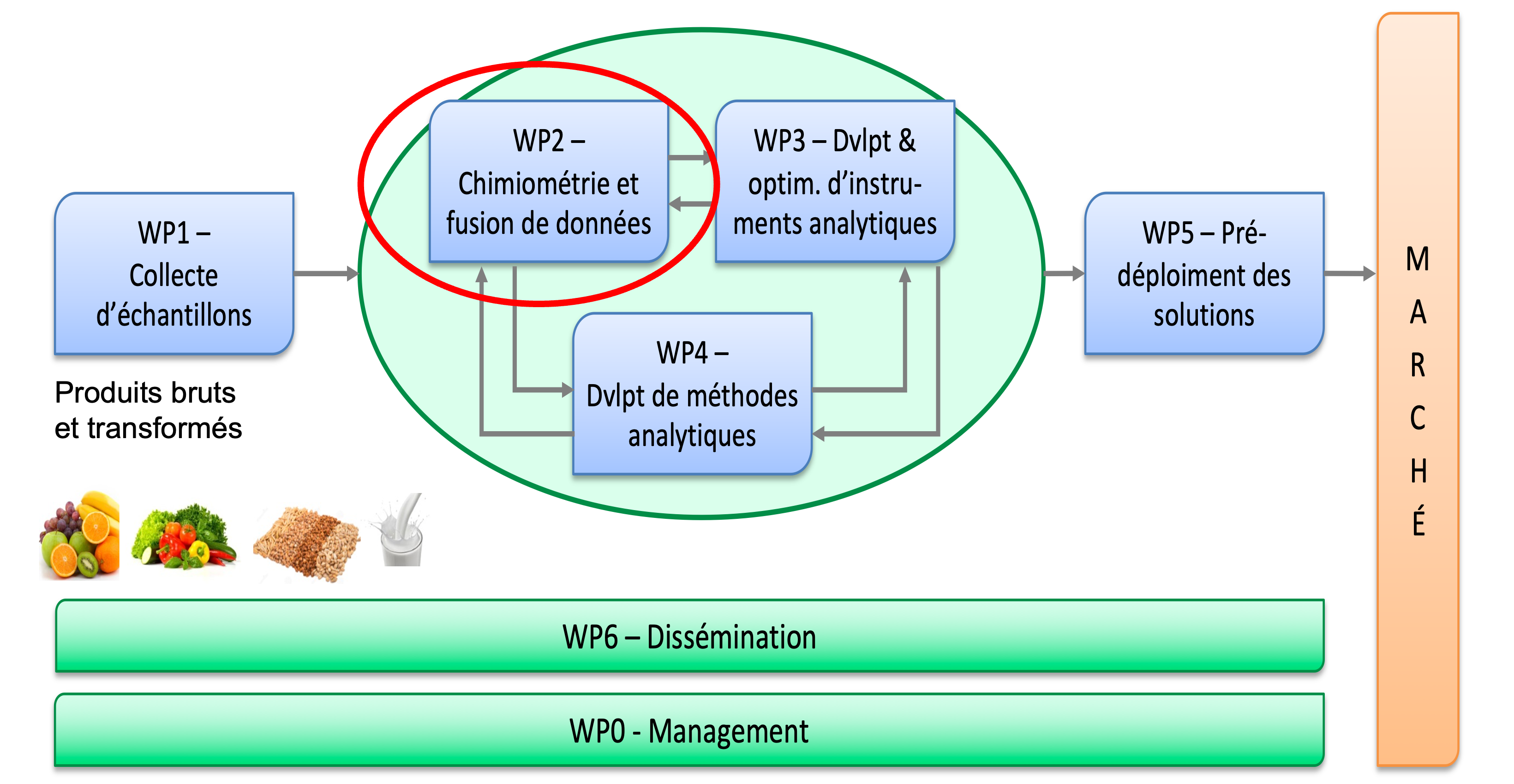
Notre objectif ici est produire et de valider un ensemble de modèles de discrimination et de classification permettant de vérifier l’authenticité des produits alimentaires analysés par une approche multi-techniques. Des algorithmes et une méthodologie de fusion de données sera spécifiquement développée pour le projet TOFoo.
Les membres du groupe en charge du WP2 sont issus d’une unité de recherche de UniLasalle (Beauvais, France), de l’université Laval (Québec, Canada) et de la société DataFrame (Nantes, France)
Apport de méthodologies de traitement des données multivariées
| Partenaire | Nom du co-responsable pour le WP2 | Expertise |
|---|---|---|
| Université Laval | Pr. Christophe B.Y. Cordella | Analyse multivariée, fusion de données Prétraitement, ontologie SAISIRⓒ Plan d'expériences Développement d'interfaces graphiques interactives Matlab Matlab, .Net |
| UniLasalle | Dr. Lyes Lakhal | Analyse multivariée, prétraitement Intelligence artificielle Matlab, Python |
| DataFrame | Dr. Dominique Bertrand | Analyse multivariée, fusion de données Prétraitement, ontologie SAISIRⓒ Matlab, C#, C |
Mise en place et adaptation du format d’enregistrement SAISIR© pour la structuration des données

Le package SAISIR© est un package de fonctions chimiométriques développé par C.B.Y. Cordella et D. Bertrand (Sté DataFrame, autre partenaire sous-traitant du projet) en 2010 et publié en 2014
Cordella, CBY, Bertrand, D. SAISIR®: a new general chemometric toolbox, Trends in Anal. Chem., 54, 2014.
Site web : http://www.chimiometrie.fr/saisir_webpage.html
SAISIR© est une ontologie simple de structuration de données qui permet un développement de code Matlab ultra rapide.
Elle se fonde sur l’utilisation primaire de 3 champs de type de données :
Notre objectif ici sera d’étendre les propriétés d’enregistrement et d’identification de SAISIR© à tous les types de données du projet TOFoo de façon à intégrer de nouvelles informations utiles à la gestion des données, à leur stockage ou à leur modélisation
Objectif de travail du WP2 pour le projet
Le WP2 a pour objectif principal de construire une séquence d’étapes (workflow) de traitement et de gestion des données pour le projet TOFoo illustrée ci-dessous.
La pierre angulaire de ce processus est l’ontologie SAISIR qui définit un cadre de programmation qui sera adapté aux nécessités du projet. L’ensemble des développements seront intégrés à une plateforme en ligne dont la structure est en développement chez l’un de nos partenaires de projet (ATOL C&D).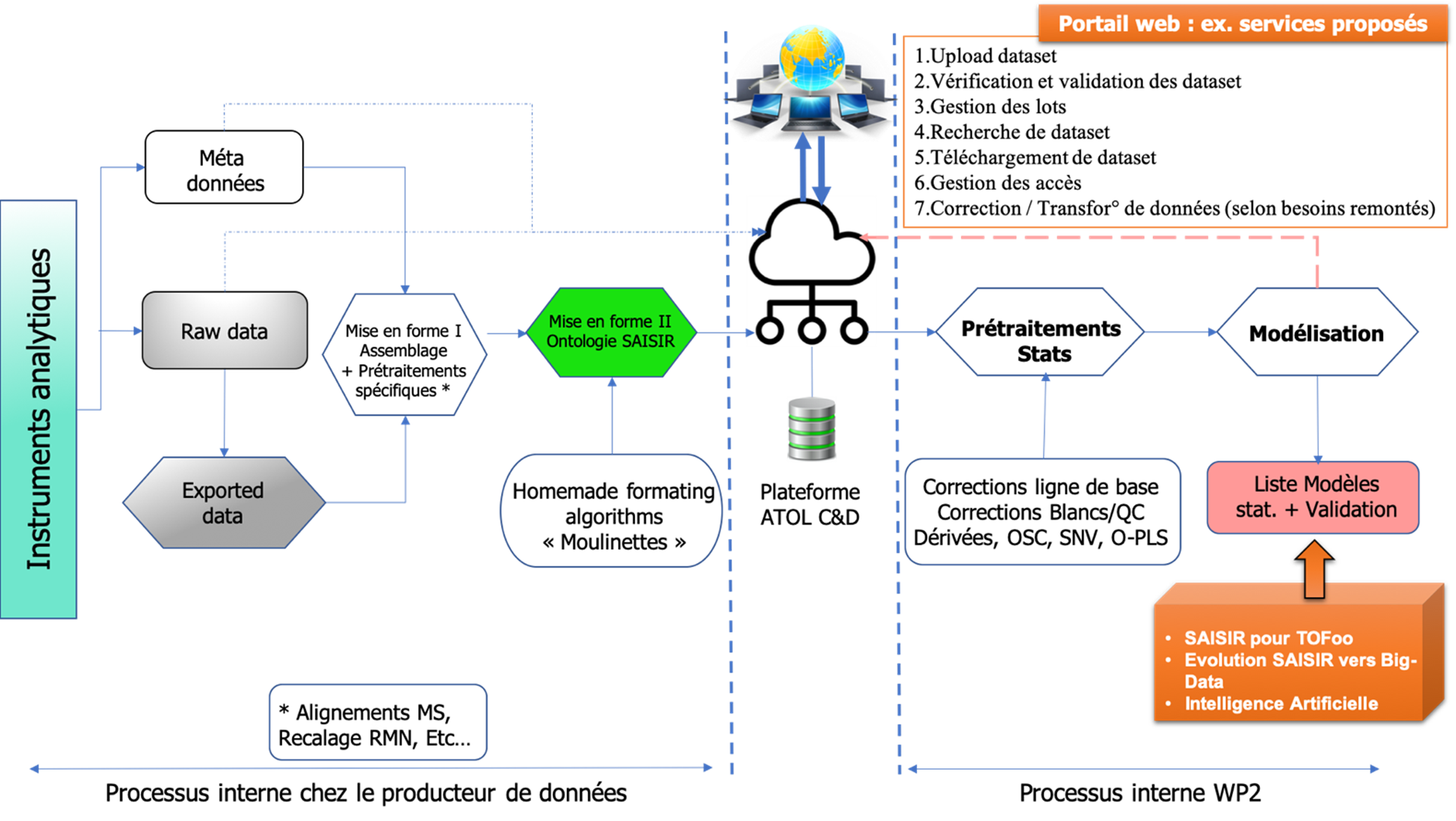
Structure SAISIR adaptée au projet TOFoo en fonction de la nature des données produites
Quelques exemples :




Légende :
.d : spectres après normalisation par le facteur ERETIC et recalés une première fois avec Icoshift (ou autre)
internalSampleNames : codes internes pour les échantillons
Scale : axe des abscisses
rawdata : spectres bruts avant tout traitement
ERETIC : pour chaque spectre, la valeur ERETIC (External Reference To access In vivo Concentration, la valeur correspond à une aire équivalente à 1 mmol/L.)
Données traitées par le projet TOFoo
LC-HRMS
GC-HRMS
Isotopie
Imagerie
Fluorescence
ICP-MS
Raman
RMN Profiling
Partenaires TOFoo

Le workpackage n°2 (WP2) du projet TOFoo vise à développer les outils de gestion et de modélisation des données multi-techniques (IRMS, RMN, HRMS et Raman). Il s’agit donc non seulement de développer les classes et méthodes assurant la mise en forme, le prétraitement et la modélisation (modèles de classification) de large ensemble de données mais également de mettre à disposition des producteurs de données une ensemble d’outils de visualisation (Histogrammes, boxplot, scatter-plots etc…).
Voici quelques exemples d’interfaces développées dans le projet et disponibles sur notre serveur web d’applications.

Statistiques Descriptives
Paramètres de représentation :

Explications du format de fichier
Le format de données pris en charge dans cette application Web est le suivant :
La 1ère colonne doit contenir les noms des échantillons (ou des observations). La 1ère ligne doit contenir les noms des variables mesurées (ou des paramètres de l’expérience) Si vous avez des colonnes non numérique comme ici dans l’exemple, placez-les en premier avant les colonnes numériques et ensuite placez vos colonnes numériques. Évitez de mettre des colonnes non-numériques à droite des colonnes numériques.
Le fichier EXCEL doit être enregistré au format .xlsx.
Output Log
Statistiques Multivariées
PCA
Paramètres de représentation :
ICA
Paramètres de représentation :
Options avancées
App Version : 1.2.5 ; Par Maxime CORDELLA ; 28/01/2023
Ete 2023
Le projet TOFoo franchit en Juin 2023 une étape décisive grâce à l'obtention de modèles d'intelligence artificielle performants sur deux matrices alimentaires : Le lait UHT et la tomate. Nos modèles sont capables de reconnaître l'origine BIO ou conventionnels des échantillons de lait UHT et de tomate avec un taux de réussite de l'ordre de 98% (après validation statistique).